Микробиота - Microbiota
Эта статья ведущий раздел не адекватно подвести итог ключевые моменты его содержания. Пожалуйста, подумайте о расширении интереса до предоставить доступный обзор обо всех важных аспектах статьи. (Октябрь 2018 г.) |
Микробиота являются "экологическими сообществами комменсальный, симбиотический и патогенный микроорганизмы "[2][3] обнаружены внутри и на всех многоклеточных организмах, изученных на сегодняшний день, от растений до животных. Микробиота включает бактерии, археи, простейшие, грибы и вирусы[нужна цитата ]. Было обнаружено, что микробиота имеет решающее значение для иммунологической, гормональной и метаболической гомеостаз своего хозяина. микробиом описывает либо коллективный геномы микроорганизмов, которые обитают в экологической нише, или самих микроорганизмов.[4][5][6]
Микробиом и хозяин появились во время эволюция как синергетический единица из эпигенетика и генетические характеристики, иногда вместе называемые холобионт.[7][8]
Вступление
Все растения и животные, из простые формы жизни для людей живут в тесной связи с микробными организмами.[9] Несколько достижений повлияли на восприятие микробиомов, в том числе:
- способность выполнять геномный анализ и анализ экспрессии генов отдельных клеток и целых микробных сообществ в дисциплинах метагеномика и метатранскриптомика[10]
- базы данных, доступные исследователям из разных дисциплин[10]
- методы математического анализа, подходящие для сложных наборов данных[10]
Биологи пришли к выводу, что микробы составляют важную часть организма. фенотип далеко за пределами случайного симбиотического тематического исследования.[10]
Типы взаимоотношений микроб-хозяин
Комменсализм, концепция, разработанная Пьер-Жозеф ван Бенеден (1809–1894), бельгийский профессор Лувенский университет в девятнадцатом веке [11] занимает центральное место в микробиоме, где микробиота колонизирует хозяина, не причиняя вреда сосуществованию. Отношения с хозяином называются мутуалистический когда организмы выполняют задачи, которые, как известно, полезны для хозяина,[12]:700[13] паразитический, когда невыгодно хозяину. Другие авторы определяют ситуацию как мутуалистическую, когда и выигрыш, и комменсальный, когда незатронутый хозяин приносит пользу симбионту.[14] Обмен питательными веществами может быть двунаправленным или однонаправленным, может зависеть от контекста и может происходить различными способами.[14] Предполагаемая микробиота, которая при нормальных обстоятельствах не вызывает заболевания, считается нормальная флора или же нормальная микробиота;[12] нормальная флора может быть не только безвредной, но и защищать хозяина.[15]
Приобретение и изменение
В первоначальное приобретение микробиоты у животных от млекопитающих до морских губки возникает при рождении и может даже происходить через линию половых клеток. У растений процесс колонизации может начинаться под землей в корневая зона вокруг прорастающего семени сперма, или происходят из надземных частей, филлосфера и цветок зона или антосфера.[16] Стабильность микробиоты ризосферы на протяжении поколений зависит от типа растений, но еще больше от состава почвы, то есть живой и нежизненной среды.[17] Клинически новая микробиота может быть получена путем трансплантации фекальной микробиоты для лечения таких инфекций, как хронические. C. difficile инфекционное заболевание.[18]
Микробиота по хозяину

Люди
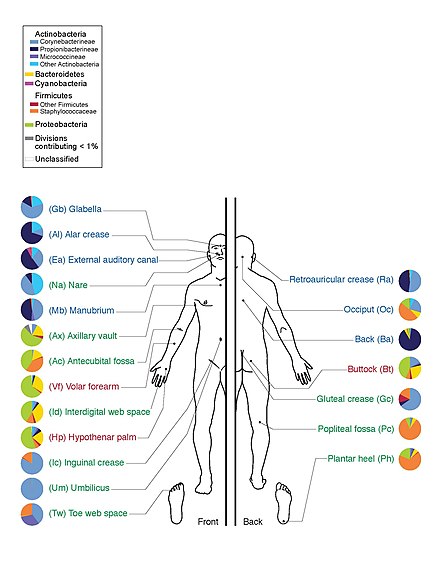
Микробиота человека включает бактерии, грибы, археи и вирусы. Микро-животные которые живут на теле человека, исключены. В человеческий микробиом относится к их геномы.[12]
Люди заселяются многими микроорганизмами; традиционная оценка заключалась в том, что люди живут с в десять раз большим количеством нечеловеческих клеток, чем человеческих клеток; более поздние оценки снизили это значение до 3: 1 и даже примерно до 1: 1.[19][20][21][22]
В Проект человеческого микробиома секвенировал геном микробиоты человека, уделяя особое внимание микробиоте, которая обычно населяет кожу, рот, нос, пищеварительный тракт и влагалище.[12] Важной вехой для него стал 2012 год, когда были опубликованы первые результаты.[23]
Животные, не относящиеся к человеку
- У земноводных есть микробиота на коже.[24] Некоторые виды способны переносить гриб, названный Batrachochytrium dendrobatidis, который у других может вызвать смертельную инфекцию Хитридиомикоз в зависимости от их микробиома, противодействие колонизации патогенами или подавление их роста антимикробными кожными пептидами.[25]
- У млекопитающих травоядные животные, такие как крупный рогатый скот, зависят от рубец микробиом для преобразования целлюлозы в белки, короткоцепочечные жирные кислоты и газы. Методы культивирования не могут предоставить информацию обо всех присутствующих микроорганизмах. Сравнительные метагеномные исследования дали удивительный результат, заключающийся в том, что отдельные виды крупного рогатого скота обладают заметно разными структурами сообщества, прогнозируемым фенотипом и метаболическим потенциалом.[26] даже несмотря на то, что их кормили идентичным рационом, содержали вместе и, по-видимому, они были функционально идентичны в использовании ресурсов клеточной стенки растений.
- мышей стали наиболее изученными млекопитающими в отношении своего микробиома. Микробиота кишечника изучалась в отношении аллергических заболеваний дыхательных путей, ожирения, желудочно-кишечных заболеваний и диабета. Перинатальное смещение микробиоты из-за низких доз антибиотиков может иметь долгосрочные последствия для будущей предрасположенности к аллергическим заболеваниям дыхательных путей. Частота появления определенных подгрупп микробов связана с серьезностью заболевания. Присутствие специфических микробов в раннем послеродовом периоде определяет будущие иммунные реакции.[27][28] Было обнаружено, что у мышей-гнотобиотов определенные кишечные бактерии передают определенный фенотип мышам-реципиентам, свободным от микробов, что способствует накоплению регуляторных Т-клеток толстой кишки, а также штаммам, которые модулируют ожирение мыши и концентрации метаболитов слепой кишки.[29] Этот комбинаторный подход позволяет на системном уровне понять вклад микробов в биологию человека.[30] Но также другие мукоидные ткани, такие как легкие и влагалище, были изучены в отношении таких заболеваний, как астма, аллергия и вагиноз.[31]
- У насекомых есть свой микробиом. Например, муравьи-листорезы образуют огромные подземные колонии, собирающие сотни килограммов листьев каждый год, и не могут напрямую переваривать целлюлозу, содержащуюся в листьях. Они поддерживают грибные сады в качестве основного источника пищи для колонии. Хотя сам гриб не переваривает целлюлозу, микробное сообщество, содержащее множество бактерий, делает это. Анализ генома микробной популяции выявил множество генов, участвующих в переваривании целлюлозы. Прогнозируемый профиль ферментов, расщепляющих углеводы, микробиома подобен таковому в рубце крупного рогатого скота, но видовой состав почти полностью отличается.[32] Микробиота кишечника плодовая муха может влиять на внешний вид кишечника, влияя на скорость обновления эпителия, расстояние между клетками и состав различных типов клеток в эпителии.[33] Когда моль Spodoptera exigua заражен бакуловирус гены, связанные с иммунитетом, подавляются, и количество кишечной микробиоты увеличивается.[34] В кишечнике двукрылых энтероэндокринный клетки воспринимают метаболиты кишечной микробиоты и координируют антибактериальные, механические и метаболические ветви врожденного иммунного ответа кишечника хозяина на комменсальную микробиоту.[35]
- У рыб есть свои микробиомы, в том числе и короткоживущие виды. Nothobranchius furzeri (бирюзовый киллифиш). Перенос кишечной микробиоты из молодых килл-рыб в килл-рыбу среднего возраста значительно увеличивает продолжительность жизни рыб-киллоев среднего возраста.[36]
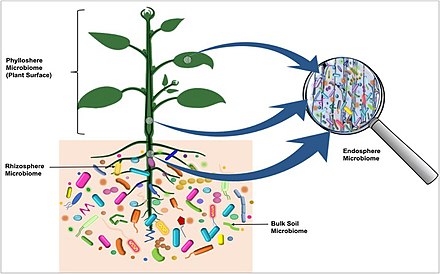
Растения

На диаграмме справа микробиота колонизирует ризосфера, проникая в корни и заселяя клубни следующего поколения через столоны, визуализируются красным цветом. Бактерии в организме матери клубень, проходящие через столоны и мигрирующие в растение, а также в клубни следующего поколения показаны синим цветом.[37]
- Почва является основным резервуаром бактерий, заселяющих клубни картофеля.
- Бактерии попадают в почву более или менее независимо от сорта картофеля.
- Бактерии могут колонизировать клубни преимущественно изнутри растений через столон.
- Бактериальная микробиота клубней картофеля состоит из бактерий, передающихся от одного поколения клубней к следующему, и бактерии, попавшие в почву, колонизируют растения картофеля через корень.[37]

Растения являются привлекательными хозяевами для микроорганизмов, поскольку они обеспечивают множество питательных веществ. Микроорганизмы на растениях могут быть эпифиты (встречается на растениях) или эндофиты (находится в тканях растений).[38][39] Оомицеты и грибы в результате конвергентной эволюции развили сходную морфологию и заняли аналогичные экологические ниши. Они развиваются гифы, нитевидные структуры, проникающие в хозяйскую клетку. В мутуалистический В таких ситуациях растение часто обменивает гексозный сахар на неорганический фосфат грибкового симбионта. Предполагается, что такие очень древние ассоциации помогли растениям, когда они впервые заселили землю.[14][40] Бактерии, способствующие росту растений (PGPB) предоставляет заводу необходимые услуги, такие как азотфиксация, солюбилизация минералов, таких как фосфор, синтез гормоны растений, прямое увеличение усвоения минералов и защита от патогенов.[41][42] PGPB могут защищать растения от патогенов, конкурируя с патогенами за экологическую нишу или субстрат, производя ингибирующие аллелохимикаты или индукция системной устойчивости растений-хозяев к патогену[16]
Исследование
Симбиотические отношения между хозяином и его микробиотой изучаются в лаборатории на предмет того, как они могут формировать иммунная система млекопитающих.[43][44] У многих животных иммунная система и микробиота могут вступать в «перекрестный диалог», обмениваясь химическими сигналами, что может позволить микробиоте влиять на иммунную реактивность и нацеливание.[45] Бактерии могут передаваться от матери к ребенку при прямом контакте и после рождение.[46] По мере становления микробиома младенца комменсальные бактерии быстро заселяют кишечник, вызывая ряд иммунных реакций и «программируя» иммунную систему на долгосрочные эффекты.[45] Бактерии способны стимулировать лимфоидную ткань, связанную со слизистой оболочкой кишечника, что позволяет ткани вырабатывать антитела к патогенам, которые могут проникать в кишечник.[45]
Микробиом человека может играть роль в активации толл-подобные рецепторы В кишечнике клетки-хозяева используют рецептор распознавания образов для распознавания опасностей и восстановления повреждений. Патогены могут влиять на это сосуществование, приводя к нарушению регуляции иммунитета, включая восприимчивость к заболеваниям, механизмы воспаление, иммунная толерантность, и аутоиммунные заболевания.[47][48]
Коэволюция микробиоты

Организмы развиваются внутри экосистем, так что изменение одного организма влияет на изменение других. В теория эволюции гологенома предполагает, что объектом естественного отбора является не отдельный организм, а организм вместе со связанными с ним организмами, включая его микробные сообщества.
коралловые рифы. Теория гологенома зародилась в исследованиях коралловых рифов.[49] Коралловые рифы - самые большие структуры, созданные живыми организмами, и содержат многочисленные и очень сложные микробные сообщества. За последние несколько десятилетий произошло резкое сокращение популяций кораллов. Изменение климата, загрязнение воды и чрезмерный вылов рыбы - три стрессовых фактора, которые были описаны как ведущие к восприимчивости к болезням. Описано более двадцати различных болезней кораллов, но лишь у немногих из них были выделены и охарактеризованы их возбудители. Обесцвечивание кораллов это самое серьезное из этих заболеваний. В Средиземном море обесцвечивание Окулина Патагоника был впервые описан в 1994 году и вскоре был определен как вызванный заражением Вибрион шилой. С 1994 по 2002 год бактериальное отбеливание О. патагоника происходило каждое лето в восточном Средиземноморье. Однако удивительно, что после 2003 г. О. патагоника в восточном Средиземноморье был устойчив к В. шилой инфекция, хотя другие болезни все же вызывают обесцвечивание. Удивление проистекает из знания о том, что кораллы живут долго, и их продолжительность жизни составляет порядка десятилетий.[50] и нет адаптивные иммунные системы.[нужна цитата ] Их врожденная иммунная система не производят антител, и, по-видимому, они не должны быть способны реагировать на новые вызовы, кроме как в эволюционных временных масштабах.[нужна цитата ]
Загадка того, как кораллам удалось приобрести устойчивость к определенному патогену, привела к предложению 2007 года о существовании динамических отношений между кораллами и их симбиотическими микробными сообществами. Считается, что, изменяя его состав, холобионт могут адаптироваться к изменяющимся условиям окружающей среды гораздо быстрее, чем только путем генетической мутации и отбора. Экстраполяция этой гипотезы на другие организмы, включая высшие растения и животных, привела к предложению теории эволюции гологенома.[49]
По состоянию на 2007 год[Обновить] теория гологенома все еще обсуждалась.[51] Серьезной критикой было утверждение, что В. шилой был ошибочно идентифицирован как возбудитель обесцвечивания кораллов, и что его присутствие в обесцвеченных О. патагоника была просто оппортунистической колонизацией.[52] Если это правда, то основное наблюдение, ведущее к теории, будет неверным. Теория приобрела значительную популярность как способ объяснения быстрых изменений в адаптации, которые иначе нельзя объяснить традиционными механизмами естественного отбора. В рамках теории хологенома холобионт стал не только основной единицей естественного отбора, но и результатом другого этапа интеграции, который также наблюдается в клетке (симбиогенез, эндосимбиоз ) и геномные уровни.[7]
Методы исследования
Целевое секвенирование ампликонов
Целевые ампликон секвенирование основывается на определенных ожиданиях относительно состава изучаемого сообщества. При секвенировании целевого ампликона филогенетически информативный маркер предназначен для секвенирования. Такой маркер должен в идеале присутствовать во всех ожидаемых организмах. Он также должен развиваться таким образом, чтобы он был достаточно консервативным, чтобы праймеры могли нацеливаться на гены от широкого круга организмов, при этом эволюционируя достаточно быстро, чтобы обеспечить более точное разрешение на таксономическом уровне. Обычным маркером исследований микробиома человека является ген бактериального 16S рРНК (т.е. «16S рДНК», последовательность ДНК, кодирующая молекулу рибосомной РНК).[53] Поскольку рибосомы присутствуют во всех живых организмах, использование 16S рДНК позволяет амплифицировать ДНК из гораздо большего числа организмов, чем при использовании другого маркера. Ген 16S рДНК содержит как медленно развивающиеся области, так и быстро развивающиеся области; первый можно использовать для конструирования широких праймеров, в то время как второй позволяет более тонкое таксономическое различие. Однако разрешение на уровне видов обычно невозможно с использованием рДНК 16S. Выбор праймера является важным этапом, поскольку все, что не может быть нацелено праймером, не будет амплифицировано и, следовательно, не будет обнаружено. Было показано, что разные наборы праймеров амплифицируют разные таксономические группы из-за вариации последовательностей.
Целевые исследования эукариотических и вирусных сообществ ограничены.[54] и при условии исключения ДНК-хозяина из амплификации и снижения эукариотической и вирусной биомассы в микробиоме человека.[55]
После секвенирования ампликонов молекулярный филогенетический методы используются для определения состава микробного сообщества. Это делается путем кластеризации ампликонов в операционные таксономические единицы (OTU) и установление филогенетических отношений между последовательностями. Из-за сложности данных такие меры расстояния, как UniFrac расстояния обычно определяются между образцами микробиома, а последующие многомерные методы выполняются на матрицах расстояний. Важным моментом является то, что масштаб данных обширен, и необходимо применять дополнительные подходы для выявления закономерностей на основе доступной информации. Инструменты, используемые для анализа данных, включают VAMPS,[56] QIIME[57] и мотылек.[58]
Метагеномное секвенирование
Метагеномика также широко используется для изучения микробных сообществ.[59][60][61] При метагеномном секвенировании ДНК извлекается непосредственно из образцов окружающей среды нецелевым образом с целью получения объективной выборки из всех генов всех членов сообщества. Недавние исследования используют дробовик Секвенирование по Сэнгеру или же пиросеквенирование для восстановления последовательностей чтения.[62] Затем считывания можно собрать в контиги. Чтобы определить филогенетическую идентичность последовательности, ее сравнивают с доступными последовательностями полного генома с использованием таких методов, как ВЗРЫВ. Одним из недостатков этого подхода является то, что многие члены микробных сообществ не имеют репрезентативного секвенированного генома, но это также относится к секвенированию ампликона 16S рРНК и является фундаментальной проблемой.[53] С секвенированием дробовика его можно решить, обеспечив высокий охват (50-100x) неизвестного генома, эффективно выполняя de novo сборка генома. Как только становится доступен полный геном неизвестного организма, его можно филогенетически сравнить, и организм будет помещен на свое место в мире. Дерево жизни, создавая новые таксоны. Возникающий подход заключается в объединении секвенирования дробовика с данными лигирования близости (Ик ) для сборки полных микробных геномов без культивирования.[63]
Несмотря на то, что метагеномика ограничена доступностью эталонных последовательностей, одним из значительных преимуществ метагеномики перед целевым секвенированием ампликонов является то, что данные метагеномики могут прояснить функциональный потенциал ДНК сообщества.[64][65] Целевые исследования генов не могут этого сделать, поскольку они только раскрывают филогенетические отношения между одним и тем же геном у разных организмов. Функциональный анализ выполняется путем сравнения восстановленных последовательностей с базами данных метагеномных аннотаций, таких как КЕГГ. Метаболические пути, в которых участвуют эти гены, затем можно предсказать с помощью таких инструментов, как MG-RAST,[66] КАМЕРА[67] и IMG / M.[68]
РНК и белковые подходы
Метатранскриптомические исследования были выполнены для изучения экспрессии генов микробных сообществ с помощью таких методов, как пиросеквенирование выделенной РНК.[69] Структурные исследования также выявили некодирующие РНК (нкРНК), такие как рибозимы из микробиоты.[70] Метапротеомика - это подход, который изучает белки, экспрессируемые микробиотой, и дает представление о ее функциональном потенциале.[71]
Проекты
В Проект человеческого микробиома запущен в 2008 году в Соединенных Штатах Национальные институты здоровья инициатива по выявлению и характеристике микроорганизмов, обнаруживаемых как у здоровых, так и у больных людей.[72] Пятилетний проект, лучше всего охарактеризованный как технико-экономическое обоснование с бюджетом в 115 миллионов долларов, проверил, как изменения в микробиоме человека связаны со здоровьем или болезнями человека.[72]
В Проект "Микробиом Земли" (EMP) - это инициатива по сбору природных образцов и анализу микробного сообщества по всему миру. Микробы очень многочисленны, разнообразны и играют важную роль в экологической системе. Еще по состоянию на 2010 год[Обновить], было подсчитано, что в результате глобальных усилий по секвенированию ДНК в окружающей среде было получено менее 1 процента общей ДНК, обнаруженной в литре морской воды или грамме почвы,[73] и конкретные взаимодействия между микробами в значительной степени неизвестны. EMP направлен на обработку до 200 000 образцов в различных биомах, создание полной базы данных микробов на Земле для характеристики окружающей среды и экосистем по микробному составу и взаимодействию. Используя эти данные, можно предложить и проверить новые экологические и эволюционные теории.[74]
Микробиота кишечника и диабет 2 типа
Микробиота кишечника очень важна для здоровья хозяина, потому что она играет роль в деградации неперевариваемых полисахаридов (ферментация резистентного крахмала, олигосахаридов, инулина), укрепляя целостность кишечника или формируя эпителий кишечника, собирая энергию, защищая от патогенов и регулируя состояние хозяина. иммунитет.[75][76]
Несколько исследований показали, что бактериальный состав кишечника у пациентов с диабетом изменился с повышением уровня Lactobacillus gasseri, Streptococcus mutans и Clostridiales с уменьшением количества бактерий, продуцирующих бутират, таких как Розбурия кишечная и Faecalibacterium prausnitzii [77][78].это изменение связано со многими факторами, такими как злоупотребление антибиотиками, диета и возраст.
Снижение продукции бутирата связано с нарушением кишечной проницаемости, этот дефект приводит к эндотоксемии, которая представляет собой повышенный уровень циркулирующих липополисахаридов из стенки грамотрицательных бактериальных клеток. Установлено, что эндотоксемия связана с развитием инсулинорезистентности.[77]
Кроме того, производство бутирата влияет на уровень серотонина.[77] Повышенный уровень серотонина способствует ожирению, которое, как известно, является фактором риска развития диабета.
Микробиоту можно пересаживать в организм человека в медицинских целях.[79]
Проблемы с конфиденциальностью
Микробная ДНК, обитающая в человеческом теле человека, может однозначно идентифицировать человека. Конфиденциальность человека может быть нарушена, если он анонимно передал данные ДНК микроба. Их состояние здоровья и личность могут быть установлены.[80][81][82]
Смотрите также
Рекомендации
- ^ Дастогеер, К.М., Тумпа, Ф.Х., Султана, А., Актер, М.А., и Чакраборти, А. (2020) «Микробиом растений - учет факторов, формирующих состав и разнообразие сообщества». Современная биология растений, 23: 100161. Дои:10.1016 / j.cpb.2020.100161.
 Материал был скопирован из этого источника, который доступен под Международная лицензия Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен под Международная лицензия Creative Commons Attribution 4.0. - ^ Ледерберг, Дж; МакКрей, AT (2001). "'Ome Sweet 'Omics - генеалогическая сокровищница слов ". Ученый. 15: 8.
- ^ Рабочая группа NIH HMP; Петерсон, Дж; Гаргес, S; и другие. (2009). "Проект микробиома человека NIH". Genome Res. 19 (12): 2317–2323. Дои:10.1101 / гр.096651.109. ЧВК 2792171. PMID 19819907.
- ^ Backhed, F .; Ley, R.E .; Sonnenburg, J. L .; Петерсон, Д. А .; Гордон, Дж. И. (2005). «Бактериальный мутуализм хозяина в кишечнике человека». Наука. 307 (5717): 1915–1920. Bibcode:2005Наука ... 307.1915B. Дои:10.1126 / science.1104816. PMID 15790844. S2CID 6332272.
- ^ Turnbaugh, P.J .; Ley, R.E .; Hamady, M .; Fraser-Liggett, C.M .; Knight, R .; Гордон, Дж. И. (2007). «Проект человеческого микробиома». Природа. 449 (7164): 804–810. Bibcode:2007Натура.449..804Т. Дои:10.1038 / природа06244. ЧВК 3709439. PMID 17943116.
- ^ Ley, R.E .; Петерсон, Д. А .; Гордон, Дж. И. (2006). «Экологические и эволюционные силы, формирующие микробное разнообразие в кишечнике человека». Клетка. 124 (4): 837–848. Дои:10.1016 / j.cell.2006.02.017. PMID 16497592. S2CID 17203181.
- ^ а б Сальвуччи, Э. (2016). «Микробиом, холобионт и сеть жизни». Критические обзоры в микробиологии. 42 (3): 485–494. Дои:10.3109 / 1040841X.2014.962478. PMID 25430522. S2CID 30677140.
- ^ Guerrero, R .; Маргулис, Линн; Берланга, М. (2013). «Симбиогенез: холобионт как единица эволюции». Международная микробиология. 16 (3): 133–43. Дои:10.2436/20.1501.01.188. PMID 24568029.
- ^ Mendes, R .; Raaijmakers, J.M. (2015). «Сходство между королевствами в функциях микробиома». Журнал ISME. 9 (9): 1905–1907. Дои:10.1038 / ismej.2015.7. ЧВК 4542044. PMID 25647346.
- ^ а б c d Bosch, T. C. G .; Макфолл-Нгай, М. Дж. (2011). «Метаорганизмы как новый рубеж». Зоология. 114 (4): 185–190. Дои:10.1016 / j.zool.2011.04.001. ЧВК 3992624. PMID 21737250.
- ^ Поро Б., Биология и сложность: история и модели комменсализма. Докторская диссертация, Лионский университет, Франция, 2014 г.
- ^ а б c d Шервуд, Линда; Уилли, Джоанна; Вулвертон, Кристофер (2013). Микробиология Прескотта (9-е изд.). Нью-Йорк: Макгроу Хилл. С. 713–721. ISBN 9780073402406. OCLC 886600661.
- ^ Куигли, Э. М. (сентябрь 2013 г.). «Кишечные бактерии в здоровье и болезни». Гастроэнтерол Гепатол (N Y). 9 (9): 560–9. ЧВК 3983973. PMID 24729765.
- ^ а б c Реми В., Тейлор Т.Н., Хасс Х, Керп Х (1994). «Везикулярная арбускулярная микориза возрастом четыреста миллионов лет». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 91 (25): 11841–3. Bibcode:1994PNAS ... 9111841R. Дои:10.1073 / пнас.91.25.11841. ЧВК 45331. PMID 11607500.
- ^ Copeland, CS (сентябрь – октябрь 2017 г.). «Мир внутри нас». Журнал здравоохранения Нового Орлеана.CS1 maint: формат даты (связь)
- ^ а б Компант С., Даффи Б., Новак Дж., Клеман С., Барка Е.А. (2005). «Использование бактерий, способствующих росту растений, для биологической борьбы с болезнями растений: принципы, механизмы действия и перспективы на будущее». Appl Environ Microbiol. 71 (9): 4951–9. Дои:10.1128 / AEM.71.9.4951-4959.2005. ЧВК 1214602. PMID 16151072.
- ^ Ткач, Анджей; Чима, Джитендер; Чандра, Говинд; Грант, Аластер; Пул, Филип С. (ноябрь 2015 г.). «Стабильность и последовательность микробиоты ризосферы зависят от типа растений и состава почвы». ISME J. 9 (11): 2349–2359. Дои:10.1038 / ismej.2015.41. ЧВК 4611498. PMID 25909975.
- ^ Copeland, CS (19 апреля 2019 г.). "Что такое Clostridium difficile?". Жизнеспособность.
- ^ Американская академия микробиологии Часто задаваемые вопросы: микробиом человека В архиве 2016-12-31 в Wayback Machine Январь 2014
- ^ Джуда Л. Рознер для журнала Microbe Magazine, февраль 2014 г. У человека в десять раз больше микробных клеток, чем клеток тела?
- ^ Элисон Эбботт для Nature News. 8 января 2016 г. Ученые опровергают миф о том, что в нашем организме больше бактерий, чем клеток человека
- ^ Отправитель, R; Fuchs, S; Milo, R (январь 2016 г.). «Действительно ли нас значительно меньше численности? Возвращаясь к соотношению бактериальных клеток и клеток-хозяев у людей». Клетка. 164 (3): 337–40. Дои:10.1016 / j.cell.2016.01.013. PMID 26824647. S2CID 1790146.
- ^ «Проект NIH Human Microbiome Project определяет нормальный бактериальный состав тела». Новости NIH. 13 июня 2012 г.
- ^ Батай, А; Ли-Круз, L; Трипати, B; Kim, H; Вальдман, Б. (январь 2016 г.). «Вариации микробиома в кожных регионах земноводных: последствия для усилий по смягчению последствий хитридиомикоза». Microb. Ecol. 71 (1): 221–32. Дои:10.1007 / s00248-015-0653-0. PMID 26271741. S2CID 12951957.
- ^ Вудхемс, округ Колумбия, Роллинз-Смит, Л.А., Алфорд, Р.А., Саймон М.А., Харрис, Р.Н. (2007). «Врожденная иммунная защита кожи амфибий: антимикробные пептиды и др.». Сохранение животных. 10 (4): 425–8. Дои:10.1111 / j.1469-1795.2007.00150.x.
- ^ Брулк Дж. М.; Антонопулос Д.А.; Miller MEB; и другие. (2009). «Геноцентрическая метагеномика микробиома рубца крупного рогатого скота, связанного с клетчаткой, выявляет кормовоспецифические гликозидгидролазы». Proc. Natl. Акад. Sci. Соединенные Штаты Америки. 106 (6): 1948–53. Bibcode:2009ПНАС..106.1948Б. Дои:10.1073 / pnas.0806191105. ЧВК 2633212. PMID 19181843.
- ^ Рассел С.Л., Gold MJ; и другие. (Май 2012 г.). «Изменения микробиоты в раннем детстве, вызванные антибиотиками, повышают восприимчивость к аллергической астме». EMBO Rep. 13 (5): 440–7. Дои:10.1038 / embor.2012.32. ЧВК 3343350. PMID 22422004.
- ^ Рассел С.Л., Голд М.Дж. и др. (Август 2014 г.). «Перинатальные изменения микробиоты кишечника, вызванные приемом антибиотиков, по-разному влияют на воспалительные заболевания легких». J Allergy Clin Immunol. 135 (1): 100–9. Дои:10.1016 / j.jaci.2014.06.027. PMID 25145536.
- ^ Turnbaugh PJ, et al. (Декабрь 2006 г.). «Микробиом кишечника, связанный с ожирением, с повышенной способностью собирать энергию». Природа. 444 (7122): 1027–31. Bibcode:2006Натура 444.1027Т. Дои:10.1038 / природа05414. PMID 17183312. S2CID 4400297.
- ^ Вера Дж. Дж., Ахерн П. П., Ридаура В. К. и др. (Январь 2014 г.). «Определение отношений фенотипа кишечного микроба-хозяина с использованием комбинаторных сообществ у мышей-гнотобиотов». Sci. Пер. Med. 6 (220): 220. Дои:10.1126 / scitranslmed.3008051. ЧВК 3973144. PMID 24452263.
- ^ Барфод, К.К .; Roggenbuck, M; Hansen, LH; Schjørring, S; Ларсен, СТ; Соренсен, SJ; Крогфельт, К.А. (2013). «Микробиом легких мыши в отношении кишечных и вагинальных бактериальных сообществ». BMC Microbiol. 13: 303. Дои:10.1186/1471-2180-13-303. ЧВК 3878784. PMID 24373613.
- ^ Суен; Скотт Джей Джей; Aylward FO; и другие. (2010). Зонненбург, Джастин (ред.). «Микробиом насекомых-травоядных с высокой способностью разлагать биомассу растений». PLOS Genet. 6 (9): e1001129. Дои:10.1371 / journal.pgen.1001129. ЧВК 2944797. PMID 20885794.
- ^ Broderick, Nichole A .; Бушон, Николас; Леметр, Бруно (2014). «Вызванные микробиотой изменения в экспрессии генов хозяина Drosophila melanogaster и морфологии кишечника». мБио. 5 (3): e01117–14. Дои:10,1128 / мБио.01117-14. ЧВК 4045073. PMID 24865556.
- ^ Якубовска, Агата К .; Фогель, Хайко; Эрреро, Сальвадор (май 2013 г.). «Увеличение микробиоты кишечника после подавления иммунитета у личинок, инфицированных бакуловирусом». PLOS Pathog. 9 (5): e1003379. Дои:10.1371 / journal.ppat.1003379. ЧВК 3662647. PMID 23717206.
- ^ Watnick, Paula I .; Джугдер, Бат-Эрдэн (01.02.2020). «Микробный контроль гомеостаза кишечника посредством передачи сигналов врожденного иммунитета энтероэндокринных клеток». Тенденции в микробиологии. 28 (2): 141–149. Дои:10.1016 / j.tim.2019.09.005. ISSN 0966-842X. ЧВК 6980660. PMID 31699645.
- ^ Тиббс Т.Н., Лопес Л.Р., Артур Дж.С. (2019). «Влияние микробиоты на развитие иммунной системы, хроническое воспаление и рак в контексте старения». Микробная клетка. 6 (8): 324–334. Дои:10.15698 / mic2019.08.685. ЧВК 6685047. PMID 31403049.
- ^ а б c Бухгольц, Ф., Антониелли, Л., Костич, Т., Сессич, А. и Миттер, Б. (2019) «Бактериальное сообщество картофеля формируется из почвы и частично наследуется из поколения в поколение». PLOS One, 14(11): e0223691. Дои:10.1371 / journal.pone.0223691.
 Материал был скопирован из этого источника, который доступен под Международная лицензия Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен под Международная лицензия Creative Commons Attribution 4.0. - ^ Берлец, Алеш (01.09.2012). «Новые методы и открытия в изучении микробиоты растений: поиск пробиотиков растений». Растениеводство. 193–194: 96–102. Дои:10.1016 / j.plantsci.2012.05.010. PMID 22794922.
- ^ Whipps, J.m .; Рука, П .; Pink, D .; Гибка, г. (2008-12-01). «Микробиология филлосферы с особым акцентом на разнообразие и генотип растений» (PDF). Журнал прикладной микробиологии. 105 (6): 1744–1755. Дои:10.1111 / j.1365-2672.2008.03906.x. ISSN 1365-2672. PMID 19120625. S2CID 35055151.
- ^ Chibucos MC, Тайлер BM (2009). «Общие темы усвоения питательных веществ растительными симбиотическими микробами, описанные в Gene Ontology». BMC Microbiology. 9 (Дополнение 1): S6. Дои:10.1186 / 1471-2180-9-S1-S6. ЧВК 2654666. PMID 19278554.
- ^ Клоппер, Дж. В (1993). «Ризобактерии, способствующие росту растений, как агенты биологической борьбы». В Меттинге, Ф. Б. мл. (Ред.). Микробная экология почвы: применение в сельском хозяйстве и управлении окружающей средой. Нью-Йорк: Marcel Dekker Inc., стр. 255–274. ISBN 978-0-8247-8737-0.
- ^ Bloemberg, G.V .; Лугтенберг, Б. Дж. Дж. (2001). «Молекулярные основы стимуляции роста растений и биоконтроля ризобактериям». Текущее мнение в области биологии растений. 4 (4): 343–350. Дои:10.1016 / S1369-5266 (00) 00183-7. PMID 11418345.
- ^ Palm, Noah W .; de Zoete, Marcel R .; Флавелл, Ричард А. (30 июня 2015 г.). «Взаимодействие иммунной и микробиоты в здоровье и болезни». Клиническая иммунология. 159 (2): 122–127. Дои:10.1016 / j.clim.2015.05.014. ISSN 1521-6616. ЧВК 4943041. PMID 26141651.
- ^ Round, июнь L .; О'Коннелл, Райан М .; Мазманян, Саркис К. (2010). «Координация толерогенных иммунных ответов комменсальной микробиотой». Журнал аутоиммунитета. 34 (3): J220 – J225. Дои:10.1016 / j.jaut.2009.11.007. ЧВК 3155383. PMID 19963349.
- ^ а б c Кахензли, Джулия; Балмер, Мария Л .; Маккой, Кэти Д. (2012). «Микробно-иммунный перекрестный разговор и регуляция иммунной системы». Иммунология. 138 (1): 12–22. Дои:10.1111 / j.1365-2567.2012.03624.x. ЧВК 3533697. PMID 22804726.
- ^ Розенберг, Юджин; Зильбер-Розенберг, Илана (2016). «Микробы управляют эволюцией животных и растений: концепция гологенома». мБио. 7 (2): e01395–15. Дои:10.1128 / mbio.01395-15. ЧВК 4817260. PMID 27034283.
- ^ Blander, J Magarian; Лонгман, Рэнди С; Илиев, Илиян Д; Зонненберг, Грегори Ф .; Артис, Дэвид (19 июля 2017 г.). «Регулирование воспаления посредством взаимодействия микробиоты с хозяином». Иммунология природы. 18 (8): 851–860. Дои:10.1038 / ni.3780. ISSN 1529-2908. ЧВК 5800875. PMID 28722709.
- ^ Никоопур, Э; Сингх, Б. (2014). «Взаимодействие микробиома и взаимодействий иммунной системы и ее влияние на болезнь и здоровье». Цели лекарств от воспаления и аллергии. 13 (2): 94–104. Дои:10.2174/1871528113666140330201056. PMID 24678760.
- ^ а б Розенберг Э., Корен О, Решеф Л., Эфрони Р., Зильбер-Розенберг И. (2007). «Роль микроорганизмов в здоровье, болезнях и эволюции кораллов». Обзоры природы Микробиология. 5 (5): 355–362. Дои:10.1038 / nrmicro1635. PMID 17384666. S2CID 2967190.
- ^ Бэрд А.Х., Бхагуоли Р., Ральф П.Дж., Такахаши С. (2009). «Обесцвечивание кораллов: роль хозяина» (PDF). Тенденции в экологии и эволюции. 24 (1): 16–20. Дои:10.1016 / j.tree.2008.09.005. PMID 19022522.
- ^ Леггат В., Эйнсворт Т., Байтелл Дж., Голубь С., Гейтс Р., Хуг-Гулдберг О., Иглесиас-Прието Р., Йеллоулис Д. (2007). «Теория хологенома игнорирует коралловый голобионт». Обзоры природы Микробиология. 5 (10): Электронная переписка. Дои:10.1038 / nrmicro1635-c1. S2CID 9031305.
- ^ Эйнсворт Т.Д., Fine M, Roff G, Hoegh-Guldberg O (2008). "Бактерии не являются основной причиной обесцвечивания средиземноморских кораллов. Oculina patagonica". Журнал ISME. 2 (1): 67–73. Дои:10.1038 / ismej.2007.88. PMID 18059488. S2CID 1032896.
- ^ а б Kuczynski, J .; Lauber, C.L .; Walters, W. A .; Парфри, Л. У .; Clemente, J.C .; Геверс, Д .; Найт, Р. (2011). «Экспериментально-аналитический инструментарий для изучения микробиома человека». Природа Обзоры Генетика. 13 (1): 47–58. Дои:10.1038 / nrg3129. ЧВК 5119550. PMID 22179717.
- ^ Марчези, Дж. Р. (2010). «Прокариотическое и эукариотическое разнообразие кишечника человека». Достижения в прикладной микробиологии Том 72. Успехи прикладной микробиологии. 72. С. 43–62. Дои:10.1016 / S0065-2164 (10) 72002-5. ISBN 9780123809896. PMID 20602987.
- ^ Vestheim, H .; Джарман, С. Н. (2008). «Блокирующие праймеры для усиления ПЦР-амплификации редких последовательностей в смешанных образцах - тематическое исследование ДНК жертвы в желудках антарктического криля». Границы зоологии. 5: 12. Дои:10.1186/1742-9994-5-12. ЧВК 2517594. PMID 18638418.
- ^ "VAMPS: Визуализация и анализ структур микробной популяции". Центр Бэй Пола, MBL, Вудс-Хоул. Получено 11 марта 2012.
- ^ Caporaso, J.G .; Kuczynski, J .; Stombaugh, J .; Биттингер, К .; Бушман, Ф. Д .; Костелло, Э. К .; Fierer, N .; Peña, A. G .; Goodrich, J. K .; Gordon, J. I .; Huttley, G.A .; Kelley, S.T .; Рыцари, D .; Koenig, J. E .; Ley, R.E .; Lozupone, C.A .; McDonald, D .; Muegge, B.D .; Pirrung, M .; Reeder, J .; Севинский, Дж. Р .; Turnbaugh, P.J .; Walters, W. A .; Widmann, J .; Яцуненко, Т .; Zaneveld, J .; Найт, Р. (2010). «QIIME позволяет анализировать данные секвенирования сообщества с высокой пропускной способностью». Методы природы. 7 (5): 335–336. Дои:10.1038 / nmeth.f.303. ЧВК 3156573. PMID 20383131.
- ^ Schloss, P.D .; Westcott, S.L .; Рябин, Т .; Hall, J. R .; Hartmann, M .; Hollister, E.B .; Лесневский, Р. А .; Oakley, B.B .; Паркс, Д. Х .; Робинсон, С. Дж .; Sahl, J. W .; Стрес, Б .; Thallinger, G.G .; Ван Хорн, Д. Дж .; Вебер, К. Ф. (2009). «Представляем mothur: программное обеспечение с открытым исходным кодом, независимое от платформы и поддерживаемое сообществом для описания и сравнения микробных сообществ». Прикладная и экологическая микробиология. 75 (23): 7537–7541. Дои:10.1128 / AEM.01541-09. ЧВК 2786419. PMID 19801464.
- ^ Turnbaugh, P.J .; Hamady, M .; Яцуненко, Т .; Cantarel, B.L .; Дункан, А .; Ley, R.E .; Согин, М.Л .; Jones, W. J .; Roe, B.A .; Affourtit, J. P .; Egholm, M .; Henrissat, B .; Heath, A.C .; Knight, R .; Гордон, Дж. И. (2008). «Микробиом ядра кишечника у тучных и худых близнецов». Природа. 457 (7228): 480–484. Bibcode:2009Натура 457..480Т. Дои:10.1038 / природа07540. ЧВК 2677729. PMID 19043404.
- ^ Qin, J .; Li, R .; Raes, J .; Arumugam, M .; Burgdorf, K. S .; Manichanh, C .; Nielsen, T .; Pons, N .; Levenez, F .; Yamada, T .; Mende, D. R .; Li, J .; Xu, J .; Li, S .; Li, D .; Cao, J .; Ван, Б .; Liang, H .; Zheng, H .; Xie, Y .; Tap, J .; Lepage, P .; Берталан, М .; Batto, J.M .; Hansen, T .; Le Paslier, D .; Linneberg, A .; Nielsen, H. B. R .; Pelletier, E .; Рено, П. (2010). «Каталог микробных генов кишечника человека, созданный с помощью метагеномного секвенирования». Природа. 464 (7285): 59–65. Bibcode:2010Натура 464 ... 59.. Дои:10.1038 / природа08821. ЧВК 3779803. PMID 20203603.
- ^ Tringe, S. G .; Фон Меринг, С .; Кобаяши, А .; Саламов, А. А .; Chen, K .; Chang, H.W .; Подар, М .; Short, J.M .; Mathur, E.J .; Detter, J.C .; Bork, P .; Hugenholtz, P .; Рубин, Э. М. (2005). «Сравнительная метагеномика микробных сообществ». Наука. 308 (5721): 554–557. Bibcode:2005Наука ... 308..554Т. CiteSeerX 10.1.1.377.2288. Дои:10.1126 / science.1107851. PMID 15845853. S2CID 161283.
- ^ Wooley, J.C .; Годзик, А .; Фридберг, И. (2010). Борн, Филип Э. (ред.). «Букварь по метагеномике». PLOS вычислительная биология. 6 (2): e1000667. Bibcode:2010PLSCB ... 6E0667W. Дои:10.1371 / journal.pcbi.1000667. ЧВК 2829047. PMID 20195499.
- ^ Уотсон, Мик; Рое, Райнер; Уокер, Алан У .; Дьюхерст, Ричард Дж .; Снеллинг, Тимоти Дж .; Иван Лячко; Лэнгфорд, Кайл В .; Press, Максимилиан О .; Мудрый, Эндрю Х. (28.02.2018). «Сборка 913 микробных геномов из метагеномного секвенирования рубца коровы». Nature Communications. 9 (1): 870. Bibcode:2018НатКо ... 9..870С. Дои:10.1038 / s41467-018-03317-6. ISSN 2041-1723. ЧВК 5830445. PMID 29491419.
- ^ Muller, J .; Szklarczyk, D .; Julien, P .; Letunic, I .; Roth, A .; Kuhn, M .; Powell, S .; Фон Меринг, С .; Doerks, T .; Дженсен, Л. Дж .; Борк, П. (2009). «EggNOG v2.0: Расширение эволюционной генеалогии генов за счет улучшенных неконтролируемых ортологичных групп, видов и функциональных аннотаций». Исследования нуклеиновых кислот. 38 (Проблема с базой данных): D190 – D195. Дои:10.1093 / нар / gkp951. ЧВК 2808932. PMID 19900971.
- ^ Канехиса, М .; Перейти к с.; Furumichi, M .; Tanabe, M .; Хиракава, М. (2009). «KEGG для представления и анализа молекулярных сетей, включающих болезни и лекарства». Исследования нуклеиновых кислот. 38 (Проблема с базой данных): D355 – D360. Дои:10.1093 / нар / gkp896. ЧВК 2808910. PMID 19880382.
- ^ Мейер, Ф .; Paarmann, D .; d'Souza, M .; Olson, R .; Glass, E.M .; Кубал, М .; Paczian, T .; Родригес, А .; Stevens, R .; Wilke, A .; Wilkening, J .; Эдвардс, Р. А. (2008). «RAST-сервер метагеномики - общедоступный ресурс для автоматического филогенетического и функционального анализа метагеномов». BMC Bioinformatics. 9: 386. Дои:10.1186/1471-2105-9-386. ЧВК 2563014. PMID 18803844.
- ^ Sun, S .; Chen, J .; Li, W .; Altintas, I .; Lin, A .; Peltier, S .; Акции, К .; Allen, E. E .; Эллисман, М .; Grethe, J .; Вули, Дж. (2010). «Киберинфраструктура сообщества для передовых исследований и анализа микробной экологии: ресурс CAMERA». Исследования нуклеиновых кислот. 39 (Проблема с базой данных): D546 – D551. Дои:10.1093 / nar / gkq1102. ЧВК 3013694. PMID 21045053.
- ^ Марковиц, В. М .; Иванова, Н. Н .; Szeto, E .; Palaniappan, K .; Чу, К .; Dalevi, D .; Чен, И. М. А .; Гречкин, Ю .; Дубчак, И .; Андерсон, I .; Lykidis, A .; Mavromatis, K .; Hugenholtz, P .; Кирпидес, Н. К. (2007). «IMG / M: система управления и анализа данных для метагеномов». Исследования нуклеиновых кислот. 36 (Проблема с базой данных): D534 – D538. Дои:10.1093 / нар / гкм869. ЧВК 2238950. PMID 17932063.
- ^ Shi, Y .; Тайсон, Г. У .; Делонг, Э. Ф. (2009). «Метатранскриптомика обнаруживает уникальные микробные малые РНК в толще воды океана». Природа. 459 (7244): 266–269. Bibcode:2009Натура.459..266S. Дои:10.1038 / природа08055. PMID 19444216. S2CID 4340144.
- ^ Jimenez, R.M .; Delwart, E .; Луптак, А (2011). «Структурный поиск выявляет рибозимы головки молотка в микробиоме человека». Журнал биологической химии. 286 (10): 7737–7743. Дои:10.1074 / jbc.C110.209288. ЧВК 3048661. PMID 21257745.
- ^ Марон, Пенсильвания; Ranjard, L .; Mougel, C .; Лемансо, П. (2007). «Метапротеомика: новый подход к изучению функциональной микробной экологии». Микробная экология. 53 (3): 486–493. Дои:10.1007 / s00248-006-9196-8. PMID 17431707. S2CID 26953155.
- ^ а б «Проект NIH Human Microbiome Project». Национальные институты здравоохранения США, Министерство здравоохранения и социальных служб, Правительство США. 2016. Архивировано с оригинал 11 июня 2016 г.. Получено 14 июн 2016.
- ^ Гилберт, Дж. А .; Мейер, Ф .; Antonopoulos, D .; и другие. (2010). "Отчет о встрече: семинар по терабазной метагеномике и видение проекта микробиома Земли". Стандарты геномных наук. 3 (3): 243–248. Дои:10.4056 / sigs.1433550. ЧВК 3035311. PMID 21304727.
- ^ Гилберт, Дж. А .; O'Dor, R .; King, N .; Фогель, Т. М. (2011). «Важность метагеномных исследований для микробной экологии: или почему Дарвин был бы метагеномным ученым». Микробная информатика и эксперименты. 1 (1): 5. Дои:10.1186/2042-5783-1-5. ЧВК 3348666. PMID 22587826.
- ^ Ибрагим, Несма (01.07.2018). "Микробиота кишечника и сахарный диабет 2 типа: в чем связь?". Афро-египетский журнал инфекционных и эндемических заболеваний. 6 (2): 112–119. Дои:10.21608 / aeji.2018.9950. ISSN 2090-7184.
- ^ Терзби, Элизабет; Джуге, Натали (2017-06-01). «Введение в микробиоту кишечника человека». Биохимический журнал. 474 (11): 1823–1836. Дои:10.1042 / BCJ20160510. ISSN 0264-6021. ЧВК 5433529. PMID 28512250.
- ^ а б c Муньос-Гарач, Арасели; Диас-Пердигонес, Кристина; Tinahones, Франсиско Дж. (Декабрь 2016 г.). «Микробиота и сахарный диабет типо 2». Endocrinología y Nutrición (на испанском). 63 (10): 560–568. Дои:10.1016 / j.endonu.2016.07.008. PMID 27633134.
- ^ Бландино, G .; Inturri, R .; Lazzara, F .; Di Rosa, M .; Малагуарнера, Л. (01.11.2016). «Влияние микробиоты кишечника на сахарный диабет». Диабет и метаболизм. 42 (5): 303–315. Дои:10.1016 / j.diabet.2016.04.004. ISSN 1262-3636. PMID 27179626.
- ^ https://microbioma.it. Цитировать журнал требует
| журнал =(помощь); Отсутствует или пусто| название =(помощь) - ^ журнал, Юэн. «Микробная ДНК в организме человека может использоваться для идентификации людей». Получено 2015-05-17.
- ^ Каллавей, Юэн (2015). «Микробиомы вызывают озабоченность в отношении конфиденциальности». Природа. 521 (7551): 136. Bibcode:2015Натура. 521..136C. Дои:10.1038 / 521136a. PMID 25971486. S2CID 4393347.
- ^ Йонг, Эд (2015-05-11). «Можно ли использовать оставленные вами микробы для вашей идентификации?». Национальная география. Получено 2015-05-17.
| Scholia имеет тема профиль для микробиом. |